CBSドメイン
| CBS domain | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
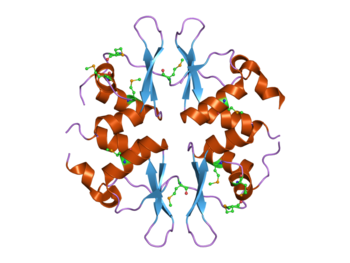 4つのCBSドメインを持つ酵母SNF4タンパク質の構造[1]。このタンパク質はAMPK複合体の一部である。 | |||||||||||
| 識別子 | |||||||||||
| 略号 | CBS | ||||||||||
| Pfam | PF00571 | ||||||||||
| InterPro | IPR000644 | ||||||||||
| SMART | CBS | ||||||||||
| PROSITE | PS51371 | ||||||||||
| SCOP | 1zfj | ||||||||||
| SUPERFAMILY | 1zfj | ||||||||||
| CDD | cd02205 | ||||||||||
| |||||||||||
CBSドメイン(英: CBS domain)は、細菌からヒトまですべての生物種でさまざまなタンパク質に存在するタンパク質ドメインである。CBSドメインは1997年に保存配列領域として初めて同定され、その名称はこのドメインを持つタンパク質の1つである、シスタチオニン-β-シンターゼに由来する[2]。CBSドメインは他にもイノシン一リン酸デヒドロゲナーゼ(IMPDH)[3]、電位依存性塩素チャネル[4][5][6][7][8]やAMP活性化プロテインキナーゼ(AMPK)[9][10]など、幅広いタンパク質に存在する。CBSドメインは、AMPやATP、S-アデノシルメチオニンなど、アデノシル基を持つ分子の結合に応答して酵素や輸送体の活性を調節する[11]。
構造
[編集]CBSドメインはβ-α-β-β-αの二次構造パターンで構成され、3本の逆並行βシートの片側に2本のαヘリックスが位置する、球状の三次構造へと折りたたまれる。CBSドメインはタンパク質配列中で常にペアで存在し、こうしたドメインのペアはβシートを介して強固な擬二量体を形成しており、CBSペアまたはBatemanドメインと呼ばれる[12][13]。CBSドメインペアはhead-to-head型(PDB: 3KPC、1PVM、2OOX)またはhead-to-tail型(PDB: 1O50、1PBJ)で結合することで円盤状のコンパクトな構造を形成し、典型的なリガンド結合領域となる溝が形成される[14][15][16][17][18]。原則として典型的結合部位の数は分子内のCBSドメインの数と一致し、CBSドメインの並び順と同じナンバリングが伝統的になされている[19]。CBSドメインはヌクレオチドのリボースと相互作用する保存されたアスパラギン酸残基を持つが、こうした溝がすべてヌクレオチドの結合などの機能を持つわけではない。また、Methanocaldococcus jannaschiiのMJ1225タンパク質では非典型的AMP結合部位も記載されているものの、その機能は今のところ不明である[20]。

リガンドの結合
[編集]CBSドメインはAMPやATP[11]、S-アデノシルメチオニン[21]などの分子のアデノシル基に結合することが示されているが、Mg2+などの金属イオンも結合する可能性がある[22][23]。こうしたさまざまなリガンドの結合に伴って、CBSドメインは関係する酵素ドメインの活性を調節する[24]。その分子機構の解明はまだ始まったばかりである[16][17][21][22][25]。現時点では、2つの異なる機構が提唱されている。1つの機構では、リガンドのヌクレオチド部分はタンパク質構造に対して本質的に何の変化も誘導せず、結合部位の静電ポテンシャルの変化がアデノシンヌクレオチドの結合の最も重要な性質であるとされる[17][26]。この「静的な」応答は、電荷による調節が有利な過程に関与しているとされる[17][26]。対照的に、2つ目の「動的な」機構は、リガンドに結合に伴うタンパク質構造の劇的なコンフォメーション変化を伴う。こうした例は、Thermus thermophilusのMg2+トランスポーターMgtEの細胞質ドメイン[22]、M. jannaschiiの機能未知タンパク質MJ0100[21][27]、Clostridium perfringensのピロホスファターゼの調節領域[28]で報告されている。
関係するドメイン
[編集]CBSドメインは、他のドメインも持つタンパク質に存在することが多い。こうしたドメインは通常、酵素活性を持つものであったり、膜輸送体やDNA結合ドメインであったりする。しかし、CBSドメインのみを持つタンパク質も、特に原核生物に多く存在する。こうしたCBSドメインのみを持つタンパク質は、キナーゼなど他のタンパク質と結合して複合体を形成し、相互作用して調節を行っている可能性がある。

疾患の原因となる変異
[編集]ヒトのCBSドメイン含有タンパク質の一部の変異は遺伝疾患の原因となる[3]。例えば、シスタチオニン-β-シンターゼの変異はホモシスチン尿症(OMIM: 236200)と呼ばれる遺伝性代謝疾患の原因となる[29]。AMPKのγサブユニットの変異はウォルフ・パーキンソン・ホワイト症候群を伴う家族性肥大型心筋症(OMIM: 600858)の原因となることが示されている。IMPDHのCBSドメインの変異は網膜色素変性症(OMIM: 180105)と呼ばれる目の疾患の原因となる。
ヒトには多数の電位依存性塩素チャネルの遺伝子が存在し、それらのいくつかのCBSドメインの変異が遺伝疾患の原因として同定されている。CLCN1の変異は筋強直症候群(OMIM: 160800)[30]、CLCN2の変異は特発性全般てんかん(OMIM: 600699)、CLCN5の変異はデント病(OMIM: 300009)、CLCN7の変異は大理石骨病(OMIM: 259700)、CLCNKBの変異はバーター症候群(OMIM: 241200)のそれぞれ原因となる。
出典
[編集]- ^ PDB: 2nye; “Structure of the Bateman2 domain of yeast Snf4: dimeric association and relevance for AMP binding”. Structure 15 (1): 65–74. (January 2007). doi:10.1016/j.str.2006.11.014. PMID 17223533.
- ^ Bateman A (January 1997). “The structure of a domain common to archaebacteria and the homocystinuria disease protein”. Trends Biochem. Sci. 22 (1): 12–3. doi:10.1016/S0968-0004(96)30046-7. PMID 9020585.
- ^ a b “CBS domains: structure, function, and pathology in human proteins”. Am. J. Physiol., Cell Physiol. 289 (6): C1369–78. (December 2005). doi:10.1152/ajpcell.00282.2005. PMID 16275737.
- ^ Ponting CP (March 1997). “CBS domains in CIC chloride channels implicated in myotonia and nephrolithiasis (kidney stones)”. J. Mol. Med. 75 (3): 160–3. PMID 9106071.
- ^ “Crystal structure of the cytoplasmic domain of the chloride channel ClC-0”. Structure 14 (2): 299–307. (February 2006). doi:10.1016/j.str.2005.10.008. PMID 16472749.
- ^ “Removal of gating in voltage-dependent ClC-2 chloride channel by point mutations affecting the pore and C-terminus CBS-2 domain”. J. Physiol. 572 (Pt 1): 173–81. (April 2006). doi:10.1113/jphysiol.2005.102392. PMC 1779660. PMID 16469788.
- ^ “The structure of the cytoplasmic domain of the chloride channel ClC-Ka reveals a conserved interaction interface”. Structure 15 (6): 715–25. (June 2007). doi:10.1016/j.str.2007.04.013. PMID 17562318.
- ^ “Nucleotide recognition by the cytoplasmic domain of the human chloride transporter ClC-5”. Nat. Struct. Mol. Biol. 14 (1): 60–7. (January 2007). doi:10.1038/nsmb1188. PMID 17195847.
- ^ “Structure of a CBS-domain pair from the regulatory gamma1 subunit of human AMPK in complex with AMP and ZMP”. Acta Crystallogr. D 63 (Pt 5): 587–96. (May 2007). doi:10.1107/S0907444907009110. PMID 17452784.
- ^ “Structure of the Bateman2 domain of yeast Snf4: dimeric association and relevance for AMP binding”. Structure 15 (1): 65–74. (January 2007). doi:10.1016/j.str.2006.11.014. PMID 17223533.
- ^ a b Kemp BE (January 2004). “Bateman domains and adenosine derivatives form a binding contract”. J. Clin. Invest. 113 (2): 182–4. doi:10.1172/JCI20846. PMC 311445. PMID 14722609.
- ^ Kemp BE (January 2004). “Bateman domains and adenosine derivatives form a binding contract”. J. Clin. Invest. 113 (2): 182–4. doi:10.1172/JCI20846. PMC 311445. PMID 14722609.
- ^ “Characteristics and crystal structure of bacterial inosine-5'-monophosphate dehydrogenase”. Biochemistry 38 (15): 4691–700. (April 1999). doi:10.1021/bi982858v. PMID 10200156.
- ^ “Structure of the Bateman2 domain of yeast Snf4: dimeric association and relevance for AMP binding”. Structure 15 (1): 65–74. (January 2007). doi:10.1016/j.str.2006.11.014. PMID 17223533.
- ^ “Nucleotide recognition by the cytoplasmic domain of the human chloride transporter ClC-5”. Nat. Struct. Mol. Biol. 14 (1): 60–7. (January 2007). doi:10.1038/nsmb1188. PMID 17195847.
- ^ a b “Crystal structure of the heterotrimer core of Saccharomyces cerevisiae AMPK homologue SNF1”. Nature 449 (7161): 492–5. (September 2007). doi:10.1038/nature06127. PMID 17851534.
- ^ a b c d “Crystal structures of the adenylate sensor from fission yeast AMP-activated protein kinase”. Science 315 (5819): 1726–9. (March 2007). doi:10.1126/science.1137503. PMID 17289942.
- ^ “Structural insight into AMPK regulation: ADP comes into play”. Structure 15 (10): 1285–95. (October 2007). doi:10.1016/j.str.2007.07.017. PMID 17937917.
- ^ “AMPK structure and regulation from three angles”. Structure 15 (10): 1161–3. (October 2007). doi:10.1016/j.str.2007.09.006. PMID 17937905.
- ^ “The Crystal Structure of Protein MJ1225 from Methanocaldococcus jannaschii Shows Strong Conservation of Key Structural Features Seen in the Eukaryal gamma-AMPK”. J Mol Biol 399 (1): 53–70. (May 2010). doi:10.1016/j.jmb.2010.03.045. PMID 20382158.
- ^ a b c “Binding of S-methyl-5'-thioadenosine and S-adenosyl-L-methionine to protein MJ0100 triggers an open-to-closed conformational change in its CBS motif pair”. J. Mol. Biol. 396 (3): 800–20. (February 2010). doi:10.1016/j.jmb.2009.12.012. PMID 20026078.
- ^ a b c “Mg2+-sensing mechanism of Mg2+ transporter MgtE probed by molecular dynamics study”. Proc. Natl. Acad. Sci. U.S.A. 105 (40): 15393–8. (October 2008). doi:10.1073/pnas.0802991105. PMC 2563093. PMID 18832160.
- ^ Hattori M, Nureki O「[Structural basis for the mechanism of Mg2 homeostasis by MgtE transporter]」『Tanpakushitsu Kakusan Koso』第53巻第3号、March 2008、242–8頁、PMID 18326297。
- ^ “CBS domains form energy-sensing modules whose binding of adenosine ligands is disrupted by disease mutations”. J. Clin. Invest. 113 (2): 274–84. (January 2004). doi:10.1172/JCI19874. PMC 311435. PMID 14722619.
- ^ “Crystal structures of the CBS and DRTGG domains of the regulatory region of Clostridiumperfringens pyrophosphatase complexed with the inhibitor, AMP, and activator, diadenosine tetraphosphate”. J. Mol. Biol. 398 (3): 400–13. (May 2010). doi:10.1016/j.jmb.2010.03.019. PMID 20303981.
- ^ a b “Structural basis for AMP binding to mammalian AMP-activated protein kinase”. Nature 449 (7161): 496–500. (September 2007). doi:10.1038/nature06161. PMID 17851531.
- ^ “Purification, crystallization and preliminary X-ray diffraction analysis of the CBS-domain pair from the Methanococcus jannaschii protein MJ0100”. Acta Crystallographica Section F 64 (Pt 10): 936–41. (October 2008). doi:10.1107/S1744309108027930. PMC 2564890. PMID 18931440.
- ^ “Crystal Structures of the CBS and DRTGG Domains of the Regulatory Region of Clostridium perfringens Pyrophosphatase Complexed with the Inhibitor, AMP, and Activator, Diadenosine Tetraphosphate”. J Mol Biol 398 (3): 400–413. (May 2010). doi:10.1016/j.jmb.2010.03.019. PMID 20303981.
- ^ “Mutations in the regulatory domain of cystathionine beta synthase can functionally suppress patient-derived mutations in cis”. Hum. Mol. Genet. 10 (6): 635–43. (March 2001). doi:10.1093/hmg/10.6.635. PMID 11230183.
- ^ Pusch M (April 2002). “Myotonia caused by mutations in the muscle chloride channel gene CLCN1”. Hum. Mutat. 19 (4): 423–34. doi:10.1002/humu.10063. PMID 11933197.
