ヘアピンリボザイム

ヘアピンリボザイム(英: hairpin ribozyme)は、リボザイムとして作用するRNAの小さな断片である。ハンマーヘッドリボザイムと同様、植物ウイルスのRNAサテライトに存在する。タバコ輪点ウイルス(TRSV)のサテライトRNAのマイナス鎖で最初に同定された。自己切断と結合(ライゲーション)反応を触媒し、ローリングサークル型のウイルス複製による産物を直鎖状または環状のサテライトRNA分子へとプロセシングする。
生物学的機能
[編集]ヘアピンリボザイムはRNAのプロセシング反応を触媒するRNAモチーフであり、自身が埋め込まれているサテライトRNA分子を複製するために必要不可欠である。この反応は自己プロセシング反応であり、分子は自身の構造の組み替えを行う。切断反応と末端結合反応はどちらもリボザイムモチーフによって媒介され、相互互換的な直鎖状と環状のサテライトRNA分子の混合物が形成される。この反応は、ローリングサークル型の複製によって形成された巨大な多量体RNA分子のプロセシングに重要である。複製サイクルの最終段階において、巨大な中間体は単位長の分子(直鎖状または環状)へとプロセシングされた後ウイルスへと詰め込まれ、他の細胞へ運ばれてそこでさらなる複製が行われる[1]。
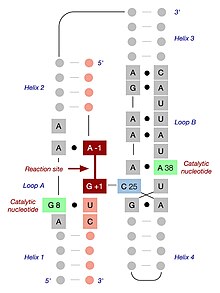
天然または人工のヘアピンリボザイム
[編集]天然のヘアピンリボザイムは3つの配列で同定されているだけである。
- タバコ輪点ウイルスのサテライトRNA(sTRSV)[3][4]
- chicory yellow mottle virusのサテライトRNA(sCYMV)[5]
- アラビスモザイクウイルスのサテライトRNA(sARMV)[6]
分子のより詳細な実験的解析を行うために、人工的に改変したより小さなヘアピンリボザイムが開発されている[7]。こうした改変は、自己プロセシング分子から無関係な部分を除去し、RNAプロセシング反応に必須な部分のみを取り出すために一般的に用いられる手法である。この過程によって、約50ヌクレオチドからなる最小触媒ドメイン(ミニマルヘアピンリボザイム)と14ヌクレオチドの基質が同定された[8]。こうした人工的配列を用いることで、複数の基質分子の切断を触媒することができる、トランスに作用するリボザイムが開発された。こうした人工的改変によって、酵素の解析に生化学的手法を用いることができるようになり、リボザイム-基質複合体の必須の構造要素が同定され、病原性ウイルスの複製の防止や個々の遺伝子の機能の研究など、生物医学分野への応用が可能な改変リボザイムの開発が行われた。
反応の化学
[編集]他のいくつかのリボザイムやタンパク質性リボヌクレアーゼと同様、ヘアピンリボザイムの切断反応は末端が2',3'-環状リン酸と5'-ヒドロキシル基となったRNA断片を作り出す。ライゲーション反応は単に切断の逆反応であるようであり、末端が2',3'-環状リン酸と5'-ヒドロキシル基のRNA断片が共有結合によって連結されて通常の3'-5'ホスホジエステル結合が形成される。
複数のリボザイムでの反応の研究により、一部のタンパク質性酵素やリボザイムとは異なり、触媒機構はRNA分子自身の作用によるものであり、金属イオンによって媒介されるものではないことが解明されている[9]。切断活性はMg2+が[Co(NH3)6]3+で置換された場合でも観察される[10]。溶液中ではCo3+はNH3と非常に強固に結合しているためNH3は解離することがなく、そのためプロトン化されることはない。このことは、金属によって触媒されるプロトン転移やRNAへの直接的な配位は存在せず、フォールディングのみに金属が必要であることを示唆している。さらに、リボザイム-阻害剤の複合体や遷移状態を模倣した状態の結晶構造からは、立体構造上A-1とG+1は離され、A-1の2'-OHは切断されるリン酸結合に対して一直線に求核攻撃を行えるよう配置されていることが示されている。さらに、G8、A38、A9は、A-1の2'-OHの脱プロトン化、リン酸の五配位型酸素の負電荷形成の安定化、G+1の5'-O離脱基のプロトン化によって触媒に関与していることが示唆されている[11][12]。
構造
[編集]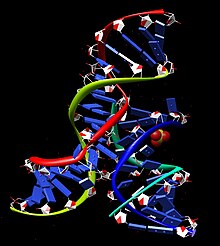
ミニマルヘアピンリボザイム-基質複合体は、2つのドメインを含む二次構造へとフォールディングする。各ドメインは、塩基対を形成した2つの短いヘリックスがインターナルループによって隔てられた構成となっている。ドメインA(ヘリックス1-ループA-ヘリックス2)には、基質とリボザイムの主要な基質認識領域が含まれる。ドメインB(ヘリックス3-ループB-ヘリックス4)はより大きく、リボザイムの主要な触媒要素が含まれる。2つのドメインはヘリックス2とヘリックス3をつなぐホスホジエステル結合によって共有結合的に連結されている。触媒反応が起こるためには、これらのドメインが互いに相互作用することが必要である[14]。
ミニマルリボザイム-基質複合体が低イオン強度条件でフォールディングした場合、2つのドメインは互いにスタッキングし、ヘアピンに似た不活性な伸びた構造を形成する[15]。触媒反応が起こるためには、2つドメインが互いに平行に並んだ、ペーパークリップに似たフォールディングを行う必要がある。さまざまな文献では、このRNAに対し「ペーパークリップ」リボザイムまたは「ヘアピン」リボザイムのいずれかの名称が付けられている。前者の名称の方がより正確であることが示されているにもかかわらず、後者の名称の方が一般的に受け入れられている。実験的には2つのドメイン間の機能的相互作用はカチオンの付加によって促進され、その正電荷は負に帯電したRNA主鎖の静電的反発を克服するのに十分である。天然のヘアピンリボザイムにおいては、2つのドメインの結合は金属イオン(Mg2+など)の結合と、2つの付加的なヘリカルドメインの存在との組み合わせによって補助される。これらの付加的ドメインはミニマルリボザイムには存在しないが、適切な三次元的フォールディングを促進する機能がある。これらのドメインはヘリックス2とヘリックス3にスタッキングし、four-way helical junctionと呼ばれる構造を形成することで2つの機能的ドメインの結合を促進する[16]。
ヘアピンリボザイムの構造と活性は広範囲にわたる相補的な実験手法によって研究がなされており、ヌクレオチドの置換、官能基の置換、コンビナトリアルセレクション(combinatorial selection)、蛍光分光法、架橋、NMR、X線結晶構造解析といった手法が用いられている。オリゴヌクレオチド合成による断片の自己集合によって機能的な複合体を形成することができるため、天然のRNAには存在しないさまざまな種類の修飾ヌクレオチドを取り込むことが可能であり、研究は促進された。こうした実験の結果、ヘアピンリボザイムがどのように基質を結合し、特定の三次元構造へフォールディングし、反応を触媒し、反応産物を放出するか、といった触媒サイクルの非常に正確な像が得られた[17]。
標的RNAの分解と抗ウイルス活性
[編集]ヘアピンリボザイムは他のRNA分子標的を切断できるように改変されている。ヘアピンリボザイムの基質特異性の大部分はヘリックス1、2内での単純なワトソン・クリック型塩基対形成によるものであるため、こうした改変が可能となっている[18]。
ヘアピンリボザイムの治療目的での開発で特に関心が持たれている領域としては、病原性ウイルスの複製の防止が挙げられる。抗ウイルス活性を持つリボザイムが作製され哺乳類細胞内での発現が行われており、さまざまな改変型リボザイムが発現細胞にHIV-1[19][20]、HBV[21]、シンドビスウイルス[22]の感染に対する耐性を付与することが示されている。
出典
[編集]- ^ Symons, RH (1997). “Plant pathogenic RNAs and RNA catalysis”. Nucleic Acids Res 25 (14): 2683–2689. doi:10.1093/nar/25.14.2683. PMC 146833. PMID 9207012.
- ^ Alam, S.; Grum-Tokars, V.; Krucinska, J.; Kundracik, M.; Wedekind, J. (2005). “Conformational heterogeneity at position U37 of an all-RNA hairpin ribozyme with implications for metal binding and the catalytic structure of the S-turn”. Biochemistry 44 (44): 14396–14408. doi:10.1021/bi051550i. PMID 16262240.
- ^ Feldstein, PA; Buzayan, JM; Bruening, G (Oct 15, 1989). “Two sequences participating in the autolytic processing of satellite tobacco ringspot virus complementary RNA.”. Gene 82 (1): 53–61. doi:10.1016/0378-1119(89)90029-2. PMID 2583519.
- ^ Hampel, A; Tritz, R (Jun 13, 1989). “RNA catalytic properties of the minimum (-)sTRSV sequence.”. Biochemistry 28 (12): 4929–4933. doi:10.1021/bi00438a002. PMID 2765519.
- ^ Rubino, L; Tousignant, ME; Steger, G; Kaper, JM (September 1990). “Nucleotide sequence and structural analysis of two satellite RNAs associated with chicory yellow mottle virus.”. The Journal of General Virology 71 (9): 1897–1903. doi:10.1099/0022-1317-71-9-1897. PMID 1698918.
- ^ Kaper, JM; Tousignant, ME; Steger, G (Jul 15, 1988). “Nucleotide sequence predicts circularity and self-cleavage of 300-ribonucleotide satellite of arabis mosaic virus.”. Biochemical and Biophysical Research Communications 154 (1): 318–325. doi:10.1016/0006-291x(88)90687-0. PMID 3395334.
- ^ Feldstein, PA; Bruening, G (1993). “Catalytically active geometry in the reversible circularization of 'mini-monomer' RNAs derived from the complementary strand of tobacco ringspot virus satellite RNA”. Nucleic Acids Research 21 (8): 1991–1998. doi:10.1093/nar/21.8.1991. PMC 309442. PMID 7684131.
- ^ Hampel, A; Tritz, R (1989). “RNA catalytic properties of the minimum (-)sTRSV sequence”. Biochemistry 28 (12): 4929–4933. doi:10.1021/bi00438a002. PMID 2765519.
- ^ Murray, J. B.; Seyhan, A. A.; Walter, N. G.; Burke, J. M.; Scott, W. G. (1998). “The hammerhead, hairpin and VS ribozymes are catalytically proficient in monovalent cations alone”. Chemistry & Biology 5 (10): 587–595. doi:10.1016/S1074-5521(98)90116-8. PMID 9818150.
- ^ Young, K. J.; Gill, F.; Grasby, J. A. (1997-10-01). “Metal Ions Play a Passive Role in the Hairpin Ribozyme Catalysed Reaction” (英語). Nucleic Acids Research 25 (19): 3760–3766. doi:10.1093/nar/25.19.3760. ISSN 0305-1048. PMC 146958. PMID 9380495.
- ^ Rupert, P. B.; Ferré-D'Amaré, A. R. (2001-04-12). “Crystal structure of a hairpin ribozyme-inhibitor complex with implications for catalysis”. Nature 410 (6830): 780–786. Bibcode: 2001Natur.410..780R. doi:10.1038/35071009. ISSN 0028-0836. PMID 11298439.
- ^ Rupert, Peter B.; Massey, Archna P.; Sigurdsson, Snorri Th; Ferré-D'Amaré, Adrian R. (2002-11-15). “Transition state stabilization by a catalytic RNA”. Science 298 (5597): 1421–1424. Bibcode: 2002Sci...298.1421R. doi:10.1126/science.1076093. ISSN 1095-9203. PMID 12376595.
- ^ Salter, J.; Krucinska, J.; Alam, S.; Grum-Tokars, V.; Wedekind, J. E. (2006). “Water in the active site of an all-RNA hairpin ribozyme and the effects of Gua8 base variants on the geometry of phosphoryl transfer”. Biochemistry 45 (3): 686–700. doi:10.1021/bi051887k. PMC 2546605. PMID 16411744.
- ^ Butcher, S. E.; Heckman, J. E.; Burke, J. M. (1995). “Reconstitution of hairpin ribozyme activity following separation of functional domains”. The Journal of Biological Chemistry 270 (50): 29648–29651. doi:10.1074/jbc.270.50.29648. PMID 8530348.
- ^ Esteban, J. A.; Walter, N. G.; Kotzorek, G.; Heckman, J. E.; Burke, J. M. (1998). “Structural basis for heterogeneous kinetics: Reengineering the hairpin ribozyme”. Proceedings of the National Academy of Sciences of the United States of America 95 (11): 6091–6096. Bibcode: 1998PNAS...95.6091E. doi:10.1073/pnas.95.11.6091. PMC 27590. PMID 9600922.
- ^ Nahas, M.; Wilson, T. J.; Ha, T.; Lilley, D. M. J. (2005). “Folding and catalysis of the hairpin ribozyme”. Biochemical Society Transactions 33 (Pt 3): 461–465. doi:10.1042/BST0330461. PMID 15916541.
- ^ Cochrane, J. C.; Strobel, S. A. (2008). “Catalytic Strategies of Self-Cleaving Ribozymes”. Accounts of Chemical Research 41 (8): 1027–1035. doi:10.1021/ar800050c. PMID 18652494.
- ^ Chowrira, BM; Burke, JM (1991). “Binding and cleavage of nucleic acids by the "hairpin" ribozyme”. Biochemistry 30 (35): 8518–8522. doi:10.1021/bi00099a003. PMID 1909564.
- ^ Yu, M; Ojwang, J; Yamada, O; Hampel, A; Rapapport, J; Looney, D; Wong-Staal, F (1993). “A hairpin ribozyme inhibits expression of diverse strains of human immunodeficiency virus type 1”. Proceedings of the National Academy of Sciences of the United States of America 90 (13): 6340–6344. Bibcode: 1993PNAS...90.6340Y. doi:10.1073/pnas.90.13.6340. PMC 46924. PMID 8327516.
- ^ “Inhibition of HIV-1 replication by RNA targeted against the LTR region.”. AIDS 19 (9): 863–870. (Jun 10, 2005). doi:10.1097/01.aids.0000171399.77500.e0. PMID 15905666.
- ^ Zu Putlitz, J.; Yu, Q.; Burke, J. M.; Wands, J. R. (1999). “Combinatorial Screening and Intracellular Antiviral Activity of Hairpin Ribozymes Directed against Hepatitis B Virus”. Journal of Virology 73 (7): 5381–5387. doi:10.1128/JVI.73.7.5381-5387.1999. PMC 112594. PMID 10364285.
- ^ Zhang, Z.; Burke, J. (2005). “Inhibition of viral replication by ribozyme: mutational analysis of the site and mechanism of antiviral activity”. Journal of Virology 79 (6): 3728–3736. doi:10.1128/JVI.79.6.3728-3736.2005. PMC 1075678. PMID 15731266.
関連文献
[編集]- Doherty, EA; Doudna JA (2001). “Ribozyme structures and mechanisms”. Annu Rev Biophys Biomol Struct 30: 457–475. doi:10.1146/annurev.biophys.30.1.457. PMID 11441810.
- Ferré-D'Amaré, AR; Rupert PB (2002). “The hairpin ribozyme: from crystal structure to function”. Biochem Soc Trans 30 (Pt 6): 1105–1109. doi:10.1042/BST0301105. PMID 12440983.
