アイソフォーム
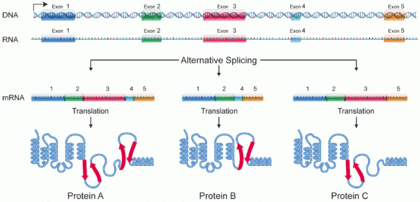
タンパク質のアイソフォーム(英: Protein isoform)またはバリアント(variant)[1]は、単一の遺伝子または遺伝子ファミリーに由来する、類似した一連のタンパク質を意味する。多くの場合、各アイソフォームは同一もしくは類似した生物学的役割を果たすが、一部のアイソフォームにのみ特有の機能が存在している場合もある。一連のアイソフォームは、選択的スプライシング、転写時に利用されるプロモーターの差異やその他の転写後修飾などによって形成されるが、翻訳後修飾によって生じた差異はアイソフォームとしては考慮されないのが一般的である。
選択的スプライシングが関与する機構では、スプライシングの過程で遺伝子上で異なるエクソン(タンパク質コード領域)が選択されたり、エクソンの異なる部分が選択されたりすることによって、1種類のmRNA前駆体から複数種類のmRNA配列が形成される。
さまざまなアイソフォーム形成機構が発見されたことで、ヒトゲノム計画で明らかにされたタンパク質コード遺伝子の数と個体に見つかるタンパク質の多様性との間の乖離を説明できるようになった。すなわち、同一の遺伝子に複数の異なるタンパク質がコードされることで、プロテオームの多様性が増大しているのである。各アイソフォームはcDNAの解析によって容易に同定することができ、ヒトの遺伝子の多くで選択的スプライシングによって複数のアイソフォームが形成されることが確認されている。各アイソフォーム間の差異はタンパク質の表面に位置する部分に生じていることが多く、ドメイン全体の欠失やループ部分の短縮などが生じていることが多い[2]。
概説
[編集]各遺伝子は、構造や組成の異なる複数種類のタンパク質を産生する能力を有している場合がある[3][4]。この過程は主にmRNAの選択的スプライシングによって調節されるが、こうした過程がヒトのプロテオームの多様性にどの程度影響を及ぼしているのかは明らかではない。というのも、mRNA転写産物レベルでの各アイソフォームの存在量とタンパク質レベルでの各アイソフォームの存在量とは必ずしも相関しているわけではないからである[5]。
翻訳された各アイソフォームの特異性は、タンパク質の構造と機能、ならびに細胞種やそれらが産生された発生段階によって決定される[3][4]。タンパク質が複数のサブユニットからなり、その各サブユニットに複数のアイソフォームが存在するときには、特異性の決定はより複雑なものとなる。
例えば、ヒトの細胞でさまざまな機能を果たすAMP活性化プロテインキナーゼ(AMPK)は3つのサブユニットからなる[6]。
- αサブユニットは触媒サブユニットで、2つのアイソフォームが存在する。α1とα2はそれぞれPRKAA1、PRKAA2遺伝子にコードされている。
- βサブユニットは調節サブユニットで、2つのアイソフォームが存在する。β1とβ2はそれぞれPRKAB1、PRKAB2遺伝子にコードされている。
- γサブユニットは調節サブユニットで、3つのアイソフォームが存在する。γ1、γ2、γ3はそれぞれPRKAG1、PRKAG2、PRKAG3遺伝子にコードされている。
ヒトの骨格筋で優先的に形成されるのはα2β2γ1であるが、肝臓で最も豊富なのはα1β2γ1である[6]。
機構
[編集]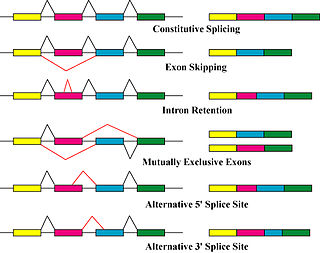
複数のアイソフォームが作り出される主要な機構は、選択的スプライシングとプロモーター利用の差異であるが、変異や多型といった遺伝的変化による修飾も異なるアイソフォームとして認識されることもある[7]。
選択的スプライシングはmRNA転写産物のアイソフォームを作り出す主要な転写後修飾過程であり、タンパク質の多様性に寄与する主要な分子機構である[4]。巨大リボヌクレオタンパク質であるスプライソソームは、核内でRNAの切断と連結を担う分子装置であり、タンパク質をコードしない領域(イントロン)を除去する[8]。
スプライシングは転写と翻訳の間に起こる過程であり、スプライシングが及ぼす主要な影響はゲノミクスの技術を用いて研究が行われてきた。例えば、マイクロアレイ解析やRNAシーケンシングが選択的スプライシングを受けた転写産物の同定やその量の測定に利用されてきた[7]。各転写産物の存在量は各アイソフォームのタンパク質レベルでの存在量の代替的な指標としてしばしば用いられるが、ゲル電気泳動や質量分析を用いたプロテオミクス実験からは、転写産物量とタンパク質量の相関はしばしば低く、通常はいずれか1つのタンパク質アイソフォームが大部分を占めていることが示されている[9]。このRNAレベルとタンパク質レベルの相違は翻訳後の段階で起こっているようであることが2015年の研究で示されているが、その機構は基本的には解明されていない[10]。そのため、選択的スプライシングによる多様性と疾患の間には重要な関連性が示唆されているものの、選択的スプライシングが新奇アイソフォームを生み出す主要な機構であるとする決定的な証拠が得られているわけではない[9]。
選択的スプライシングは一般的には緊密に制御された過程であり、選択的転写産物はスプライシング装置によって意図的に作り出される。しかしながら、このような転写産物はノイジースプライシング(noisy splicing)と呼ばれるスプライシングのエラーによっても作り出されることがあり、それらもタンパク質アイソフォームへと翻訳される可能性がある。複数のエクソンを持つ遺伝子の約95%が選択的スプライシングを受けると考えられているが、ノイジースプライシングに関するある研究では低存在量の転写産物の大部分はノイズであることが観察されており、細胞内に存在する選択的転写産物やタンパク質アイソフォームの大部分は機能的でないことが予測されている[11]。
他の転写制御・転写後制御段階によってもさまざまなタンパク質アイソフォームが作り出される[12]。細胞の転写装置(RNAポリメラーゼ、転写因子や他の酵素)が異なるプロモーターから転写を開始することによって、わずかに異なる転写産物とタンパク質アイソフォームが作り出される。
特徴
[編集]一般的に、ある遺伝子から生み出されるタンパク質アイソフォームのうちの1つが、その普遍性や他の種のオルソログ(または機能的アナログ)との配列類似性などの基準によって、標準的な配列としてラベルされる[13]。各アイソフォームは、そのほとんどが類似した配列を有していたり、一部または大部分のエクソンを標準的配列と共有していたりするため、類似した機能を有すると推測される。しかし、一部のアイソフォームにはかなり大きな差異が存在し(例えばトランススプライシングなどの機構によって)、標準的配列とほとんどまたは全くエクソンを共有していないこともある。加えて、それらは異なる生物学的影響を及ぼすこともあり、例えば極端な場合として、あるアイソフォームは細胞の生存を促進し、一方で他のアイソフォームは細胞死を促進する場合もある。また、基本的な機能は類似しているが、細胞内局在が異なることもある[14]。2016年の研究では、1492の遺伝子の全てのアイソフォームが機能的に特徴づけられ、大部分のアイソフォームが機能的に異なる"functional alloform"として振る舞うことが示された。この研究の著者らは、アイソフォームの大部分の機能が重複していないという観察をもとに、各アイソフォームは異なるタンパク質のように振る舞うという結論に達した[15]。一方で、各アイソフォームの機能は一般的には個別に決定される必要があり、同定または予測されたアイソフォームの大部分は機能未知のままである。
関連する概念
[編集]グリコフォーム
[編集]グリコフォームもしくはグライコフォーム(glycoform)は、付加された糖鎖の数や種類だけが異なるタンパク質である。糖タンパク質はしばしば、付加された糖類やオリゴ糖が異なる多数のグリコフォームから構成される。こうした差異は、グリコシル化の過程の生合成の差異や、グリコシダーゼやグリコシルトランスフェラーゼの作用の差異によって生じる。グリコフォームは詳細な化学的分析によっても検出することができるが、レクチンアフィニティークロマトグラフィーやレクチンアフィニティー電気泳動など、レクチンに対する反応の差を利用した簡便な検出も可能である。グリコフォームが存在する糖タンパク質の例としては、オロソムコイド、アンチトリプシン、ハプトグロビンなどの血漿タンパク質が挙げられる。NCAMには、ポリシアル酸からなる、一般的でない糖鎖修飾を持つグリコフォームがみられる。
例
[編集]- アクチン: 保存されたタンパク質であるにもかかわらず、さまざまな(哺乳類では少なくとも6種類の)アイソフォームが存在する。
- クレアチンキナーゼ: 血中に存在し、心筋梗塞の診断に利用される。3つのアイソフォームが存在する。
- ヒアルロン酸合成酵素: ヒアルロン酸の合成を担う。哺乳類細胞では3つのアイソフォームが存在する。
- グルクロン酸転移酵素: 多くの薬剤や環境汚染物質、有毒な内在化合物の解毒を担う酵素のスーパーファミリー。ヒトゲノムは16種類のアイソフォームがコードされていることが知られている[16]。
- モノアミン酸化酵素: モノアミンの酸化を触媒する酵素のファミリーで、MAO-AとMAO-Bの2つのアイソフォームが存在する。
出典
[編集]- ^ “Alternative splicing and genome complexity”. Nature Genetics 30 (1): 29–30. (January 2002). doi:10.1038/ng803. PMID 11743582.
- ^ Kozlowski, L.; Orlowski, J.; Bujnicki, J. M. (2012). “Structure Prediction for Alternatively Spliced Proteins”. Alternative pre-mRNA Splicing. pp. 582. doi:10.1002/9783527636778.ch54. ISBN 9783527636778
- ^ a b “Generation of protein isoform diversity by alternative splicing: mechanistic and biological implications”. Annual Review of Cell Biology 3 (1): 207–42. (1987-01-01). doi:10.1146/annurev.cb.03.110187.001231. PMID 2891362.
- ^ a b c “Alternative splicing: a ubiquitous mechanism for the generation of multiple protein isoforms from single genes”. Annual Review of Biochemistry 56 (1): 467–95. (1987-01-01). doi:10.1146/annurev.bi.56.070187.002343. PMID 3304142.
- ^ “On the Dependency of Cellular Protein Levels on mRNA Abundance”. Cell 165 (3): 535–50. (April 2016). doi:10.1016/j.cell.2016.03.014. PMID 27104977.
- ^ a b “Evolving Lessons on the Complex Role of AMPK in Normal Physiology and Cancer” (English). Trends in Pharmacological Sciences 37 (3): 192–206. (March 2016). doi:10.1016/j.tips.2015.11.007. PMC 4764394. PMID 26711141.
- ^ a b “Alternative splicing: a pivotal step between eukaryotic transcription and translation” (英語). Nature Reviews. Molecular Cell Biology 14 (3): 153–65. (March 2013). doi:10.1038/nrm3525. PMID 23385723.
- ^ “Mechanisms and Regulation of Alternative Pre-mRNA Splicing”. Annual Review of Biochemistry 84 (1): 291–323. (2015-01-01). doi:10.1146/annurev-biochem-060614-034316. PMC 4526142. PMID 25784052.
- ^ a b “Alternative Splicing May Not Be the Key to Proteome Complexity”. Trends in Biochemical Sciences 42 (2): 98–110. (February 2017). doi:10.1016/j.tibs.2016.08.008. PMID 27712956.
- ^ “Genomic variation. Impact of regulatory variation from RNA to protein”. Science 347 (6222): 664–7. (February 2015). doi:10.1126/science.1260793. PMC 4507520. PMID 25657249.
- ^ “Noisy splicing drives mRNA isoform diversity in human cells”. PLoS Genetics 6 (12): e1001236. (December 2010). doi:10.1371/journal.pgen.1001236. PMC 3000347. PMID 21151575.
- ^ “Proteoform: a single term describing protein complexity” (英語). Nature Methods 10 (3): 186–7. (March 2013). doi:10.1038/nmeth.2369. PMC 4114032. PMID 23443629.
- ^ “Revisiting the identification of canonical splice isoforms through integration of functional genomics and proteomics evidence”. Proteomics 14 (23-24): 2709–18. (December 2014). doi:10.1002/pmic.201400170. PMC 4372202. PMID 25265570.
- ^ “Cell death or survival promoted by alternative isoforms of ErbB4”. Molecular Biology of the Cell 21 (23): 4275–86. (December 2010). doi:10.1091/mbc.E10-04-0332. PMC 2993754. PMID 20943952.
- ^ “Widespread Expansion of Protein Interaction Capabilities by Alternative Splicing”. Cell 164 (4): 805–17. (February 2016). doi:10.1016/j.cell.2016.01.029. PMC 4882190. PMID 26871637.
- ^ “Substrate specificity of the human UDP-glucuronosyltransferase UGT2B4 and UGT2B7. Identification of a critical aromatic amino acid residue at position 33”. The FEBS Journal 274 (5): 1256–64. (March 2007). doi:10.1111/j.1742-4658.2007.05670.x. PMID 17263731.
